🗎 Анализ научной статьи
Содержание
🗎 Анализ научной статьи¶
Сдайте отчет с описанием статьи в виде блокнота ipynb или текстового документа (Word). В отчете Вы должны продемонстрировать умение создания структурированного текста со ссылками и нумерованными списками. Можно вместе с отчетом прикрепить полный текст статьи, особенно если ее трудно найти в открытом доступе. Оценка включает устное сообщение на семинаре в группе.
Требования к статье¶
Необходимо найти статью на интересную тему по специальности, ознакомиться с содержанием (прочитать) и извлечь признаки для описания проведенного в работе анализа данных. Ожидается, что это свежая экспериментальная статья с участием животных на английском языке в открытом журнале с доступом к исходным данным. Допускается статья в русскоязычном журнале при проблемах с английским языком. Допускается обзорная статья, если в ней проводился статистический мета-анализ.
Формальные требования:
Полная статья в профильном журнале по биологии (медицине) выпуска за последние три года или препринт.
Желательно открытый доступ к данным.
Этапы описания:
Выходные данные статьи (ссылка, квартиль по Scopus) (см. Анализ публикационного ландшафта)
Объект исследования (вид животного);
Измеряемые зависимые параметры (приборы, единицы, предобработка);
Повторы, объем выборки (ограничения, контроль);
Факторы (уровни сравнения);
Методы статистики (для описания можно выбрать один, если в работе несколько разных анализов);
Что нужно, чтобы повторить? Найти соответствующий метод в пакете scipy.stats или statsmodels или описать специальный модуль для нестандартного метода.
Пример¶
Gut microbiota dynamics in Parkinsonian mice (Динамика микробиоты кишечника у мышей с паркинсонизмом)
журнал: ACS Chemical Neuroscience
адрес: https://pubs.acs.org/doi/abs/10.1021/acschemneuro.0c00386 (полный текст)
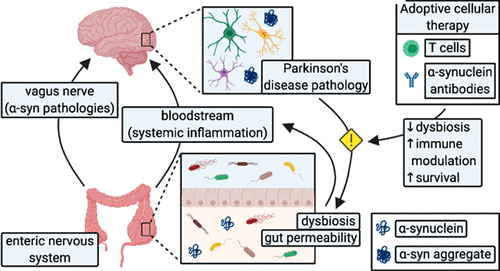
Рис. 13 Схема развития болезни Паркинсона¶
объект: микробиота кишечника мыши
субъект: особь мыши M83 (склонность к накоплению альфа-синуклеина + введение α-syn)
N: 17. 10 повторов терапии, остальное - контроль.
образцы: фекалии, возобновляемость ~ 1 час. (57 шт.), но брали раз в 1.5 месяца. (ограниченный доступ к сложному анализу?)
измеряемые показатели: образцы 16S рибосомальной РНК (1 115 уникальных OTU из 1,340,271 ридов)
предобработка: стандартные методы при амплификации в пакете QIIME2 на Python.
расчетные параметры: филогенетическая дистанция UniFrac (разработан в 2005) в пакете Phyloseq на R.
факторы: воздействие (терапия, контроль (соляной раствор)), время после инъекции (0, 6, 12 недель)
гипотеза: разный кластеринг таксономических групп бактерий в динамике болезни/лечения.
методы статистики:
непарный U-тест Манна-Уитни и корректировка с использованием метода Бенджамини-Хохберга - для оценки различия в показателях альфа-разнообразия между группами в каждый момент времени;
тест Вальда (Wald’s) - для идентификации тех ОТЮ, которые были значительно отличались по обильности ОТЮ между группами в каждый момент времени;
Permutational multivariate analysis of variance (PERMANOVA) из пакета vegan на R - для оценки различий в невзвешенных измерениях дистанции UniFrac между группами с течением времени.
Чтобы повторить:
виварий с данной линией мышей.
средства для клеточной терапии.
контейнеры для доставки кала в лабораторию.
амплификация.
пакет анализа QIIME2 (знание языка Python)
RStudio с пакетом расчета UniFrac (знание языка R)
Если способы анализа Вас заинтересовали, то оцените доступность использованных в статье методов анализа в программных пакетах на Python. Если найти готовые реализации не удалось, приведите ссылки на формальное описание метода (статья в Википедии, оригинальная статья авторов метода).
Тест Манна-Уитни доступен в пакете
scipy.stats.
scipy.stats.mannwhitneyu(x, y, use_continuity=True, alternative=None)
#Compute the Mann-Whitney rank test on samples x and y.
Метод
results.wald_test()встроен в OLS в statsmodels.PERMANOVAдоступна в пакете skbioskbio.stats.distance.permanova()или через вызов функции Adonis https://rdrr.io/rforge/vegan/man/adonis.html
Пример¶
https://www.elibrary.ru/item.asp?id=43938827

Рис. 14 Выходные данные статьи на сайте РИНЦ¶
объект: воздействие препаратов на кровь и клещей
субъект: особь кошки.
образцы: 36 кровь в начале и конце лечения, 3 группы по 6 животных в каждой группе.
измеряемые показатели: наличие/отсутствие клещей в соскобе, гематологические показатели
предобработка: стандартный общий и биохимический анализ крови
расчетные параметры: -
факторы: методы лечения 3 шт.
гипотеза: препараты действуют одинаково.
методы статистики: критерий Стьюдента
Чтобы повторить:
лечить котов с ушными клещами разными препаратами
брать на анализ кровь и соскоб в начале и конце лечения
сравнить средние по выборкам тестом Стьюдента
См.также
При возникновении сложности с получением полнотекстовой версии, попробуйте поискать копию статьи на других сайтах.
Актуальный адрес сайта sci-hub для доступа к полным текстам можно узнать в https://vk.com/sci_hub
