Сведенья о пациентах
Содержание
Сведенья о пациентах¶
Представим ситуацию, что вам прислали файл с данными о результатах обследования группы пациентов. О формате файла известно, что он текстовый. Допустим, что Вам нужны лишь сведения о поле и возрасте пациентов, чтобы принять решение о дальнейших шагах.
ssylka = 'd/data.txt'
Pandas предоставляет методы для чтения не совсем идеальных файлов данных, с которыми приходится сталкиваться.
Попробуем справиться с особенностями данного файла, которые могут помешать нам загрузить данные, если мы не будем задавать никаких дополнительных параметров к методу чтения текстовых файлов наиболее распространённого формата - CSV (comma-separated values).
D = pd.read_csv(ssylka)
D.head()
| # Data from the article | |
|---|---|
| 0 | # Schleiger E. и др. Poststroke QEEG informs e... |
| 1 | # http://onlinelibrary.wiley.com/doi/10.1111/p... |
| 2 | ID\tHemisphere\tAge\tGender\tMoCA predischarge... |
| 3 | 1\tR\t77\tM\t26.0\t26.0\t3\t1.0\t1\t1 |
| 4 | 2L\tL\t84\tM\t15.0\t21.0\t1\t0.0\t1\t0 |
Содержимое файла загружено в таблицу, однако не так, как нам хотелось бы.
Первые строчки, начинающиеся со знака # похожи на комментарии. Остальные строчки содержат наклонную черту с буквой t - обозначение символа табуляции (длинного пробела фиксированной ширины для формирования таблиц).
D = pd.read_csv(ssylka, delimiter='\t', comment='#')
D.head()
| ID | Hemisphere | Age | Gender | MoCA predischarge | MoCA outcome | NIHSS predischarge | NIHSS outcome | mRS predischarge | mRS outcome | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | R | 77 | M | 26.0 | 26.0 | 3 | 1.0 | 1 | 1 |
| 1 | 2L | L | 84 | M | 15.0 | 21.0 | 1 | 0.0 | 1 | 0 |
| 2 | 3 | L | 47 | M | 13.0 | 22.0 | 3 | 0.0 | 2 | 0 |
| 3 | 4L | R | 72 | M | 20.0 | 23.0 | 10 | 1.0 | 4 | 1 |
| 4 | 5 | L | 61 | F | 26.0 | NaN | 0 | NaN | 0 | 0 |
С помощью дополнительных параметров мы указали команде чтения, что нужно пропускать комментарии и использовать специальный разделитель.
Однако, в таблице много колонок, а нам нужны только две. При работе с большими наборами данных, где колонок могут быть сотни, может быть существенным ограничить количество загружаемых колонок. Для этого воспользуемся соответствующим параметром.
D = pd.read_csv(ssylka, delimiter='\t', comment='#', usecols=['Age','Gender'])
D
| Age | Gender | |
|---|---|---|
| 0 | 77 | M |
| 1 | 84 | M |
| 2 | 47 | M |
| 3 | 72 | M |
| 4 | 61 | F |
| 5 | 78 | F |
| 6 | 49 | M |
| 7 | 79 | F |
| 8 | 77 | M |
| 9 | 74 | M |
| 10 | 54 | M |
| 11 | 65 | M |
| 12 | 80 | M |
| 13 | 87 | F |
| 14 | 76 | M |
| 15 | 18 | F |
| 16 | 61 | M |
| 17 | 72 | F |
| 18 | 52 | M |
| 19 | 66 | M |
| 20 | 67 | M |
| 21 | 71 | F |
| 22 | 61 | F |
| 23 | 78 | F |
| 24 | 69 | F |
| 25 | 74 | M |
| 26 | 66 | M |
| 27 | 67 | M |
| 28 | 81 | F |
| 29 | 46 | M |
| 30 | 69 | M |
| 31 | 53 | M |
| 32 | 57 | M |
| 33 | 72 | F |
| 34 | 69 | M |
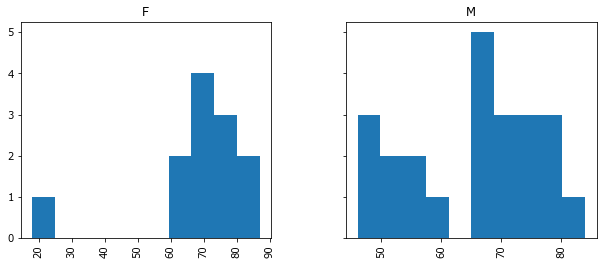
По этим колонкам можно оценить половозрастной состав выборки пациентов.
D.Gender.value_counts()
M 23
F 12
Name: Gender, dtype: int64
D.hist(by='Gender', sharey=True, figsize=(10,4));
Рассмотрим этот набор данных, описывающий пациентов, принявших участие в исследовании Schleiger E. et al. Poststroke QEEG informs early prognostication of cognitive impairment //Psychophysiology. – 2016.
Задача исследования было найти биомаркер когнитивных нарушений после инсульта.
Представим, что мы нашли онлайн версию статьи. Как извлечь данные из веб-страницы?
Table 1. Patient Demographics
| ID | Hemisphere | Age | Gender | MoCA predischarge | MoCA outcome | NIHSS predischarge | NIHSS outcome | mRS predischarge | mRS outcome |
|---|---|---|---|---|---|---|---|---|---|
| |||||||||
| 1 | R | 77 | M | 26 | 26 | 3 | 1 | 1 | 1 |
| 2L | L | 84 | M | 15 | 21 | 1 | 0 | 1 | 0 |
| 3 | L | 47 | M | 13 | 22 | 3 | 0 | 2 | 0 |
| 4L | R | 72 | M | 20 | 23 | 10 | 1 | 4 | 1 |
| 5 | L | 61 | F | 26 | NA | 0 | NA | 0 | 0 |
| 6W | L | 78 | F | 25 | 24 | 0 | 0 | 0 | 0 |
| 7 | L | 49 | M | NA | 27 | 5 | 1 | 2 | 2 |
| 8L | L | 79 | F | NA | 14 | 7 | 4 | 4 | 4 |
| 9L | R | 77 | M | NA | 15 | 15 | 7 | 5 | 4 |
| 10 | L | 74 | M | NA | 22 | 3 | 0 | 1 | 2 |
| 11WL | L | 54 | M | 28 | 30 | 0 | 0 | 0 | 0 |
| 12 | L | 65 | M | 26 | 29 | 1 | 0 | 1 | 0 |
| 13 | R | 80 | M | NA | NA | 3 | 0 | 2 | 1 |
| 14W | L | 87 | F | 16 | NA | 5 | NA | 4 | 3 |
| 15 | L | 76 | M | NA | NA | 4 | NA | 3 | 3 |
| 16 | R | 18 | F | 28 | 30 | 0 | 0 | 1 | 1 |
| 17 | L | 61 | M | 24 | 27 | 0 | 0 | 2 | 0 |
| 18 | R | 72 | F | 21 | 21 | 1 | 0 | 1 | 2 |
| 19L | R | 52 | M | NA | 27 | 21 | 13 | 5 | 4 |
| 20L | R | 66 | M | 22 | 28 | 7 | 3 | 4 | 3 |
| 21 | R | 67 | M | 18 | 24 | 5 | 1 | 3 | 2 |
| 22W | R | 71 | F | 11 | 25 | 2 | 1 | 3 | 0 |
| 23W | R | 61 | F | 25 | 28 | 4 | 1 | 3 | 1 |
| 24W | R | 78 | F | 15 | 20 | 19 | 7 | 5 | 3 |
| 25 | R | 69 | F | 27 | 27 | 0 | 1 | 1 | 1 |
| 26 | R | 74 | M | 23 | 25 | 0 | 1 | 1 | 2 |
| 27 | R | 66 | M | 23 | 23 | 0 | 0 | 0 | 1 |
| 28W | L | 67 | M | 28 | 29 | 0 | 0 | 0 | 0 |
| 29W | R | 81 | F | 18 | 23 | 1 | 0 | 2 | 1 |
| 30 | R | 46 | M | 29 | 29 | 0 | 0 | 0 | 0 |
| 31W | R | 69 | M | 24 | 27 | 1 | 0 | 1 | 0 |
| 32 | L | 53 | M | NA | 27 | 0 | 0 | 1 | 1 |
| 33W | R | 57 | M | 26 | 26 | 1 | 0 | 1 | 0 |
| 34WL | R | 72 | F | 15 | 14 | 2 | 1 | 3 | 2 |
| 35 | R | 69 | M | 26 | 27 | 0 | 0 | 2 | 0 |
Выделяем/копируем текст таблицы, делаем из него длинную строку и через строковый буфер (StringIO) передаём в функцию чтения табличных файлов как будто это текстовый файл стандартного табличного формата.
D=pd.read_table(StringIO('''ID Hemisphere Age Gender MoCA predischarge MoCA outcome NIHSS predischarge NIHSS outcome mRS predischarge mRS outcome
1 R 77 M 26 26 3 1 1 1
2L L 84 M 15 21 1 0 1 0
3 L 47 M 13 22 3 0 2 0
4L R 72 M 20 23 10 1 4 1
5 L 61 F 26 NA 0 NA 0 0
6W L 78 F 25 24 0 0 0 0
7 L 49 M NA 27 5 1 2 2
8L L 79 F NA 14 7 4 4 4
9L R 77 M NA 15 15 7 5 4
10 L 74 M NA 22 3 0 1 2
11WL L 54 M 28 30 0 0 0 0
12 L 65 M 26 29 1 0 1 0
13 R 80 M NA NA 3 0 2 1
14W L 87 F 16 NA 5 NA 4 3
15 L 76 M NA NA 4 NA 3 3
16 R 18 F 28 30 0 0 1 1
17 L 61 M 24 27 0 0 2 0
18 R 72 F 21 21 1 0 1 2
19L R 52 M NA 27 21 13 5 4
20L R 66 M 22 28 7 3 4 3
21 R 67 M 18 24 5 1 3 2
22W R 71 F 11 25 2 1 3 0
23W R 61 F 25 28 4 1 3 1
24W R 78 F 15 20 19 7 5 3
25 R 69 F 27 27 0 1 1 1
26 R 74 M 23 25 0 1 1 2
27 R 66 M 23 23 0 0 0 1
28W L 67 M 28 29 0 0 0 0
29W R 81 F 18 23 1 0 2 1
30 R 46 M 29 29 0 0 0 0
31W R 69 M 24 27 1 0 1 0
32 L 53 M NA 27 0 0 1 1
33W R 57 M 26 26 1 0 1 0
34WL R 72 F 15 14 2 1 3 2
35 R 69 M 26 27 0 0 2 0'''))
D
| ID | Hemisphere | Age | Gender | MoCA predischarge | MoCA outcome | NIHSS predischarge | NIHSS outcome | mRS predischarge | mRS outcome | |
|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | R | 77 | M | 26.0 | 26.0 | 3 | 1.0 | 1 | 1 |
| 1 | 2L | L | 84 | M | 15.0 | 21.0 | 1 | 0.0 | 1 | 0 |
| 2 | 3 | L | 47 | M | 13.0 | 22.0 | 3 | 0.0 | 2 | 0 |
| 3 | 4L | R | 72 | M | 20.0 | 23.0 | 10 | 1.0 | 4 | 1 |
| 4 | 5 | L | 61 | F | 26.0 | NaN | 0 | NaN | 0 | 0 |
| 5 | 6W | L | 78 | F | 25.0 | 24.0 | 0 | 0.0 | 0 | 0 |
| 6 | 7 | L | 49 | M | NaN | 27.0 | 5 | 1.0 | 2 | 2 |
| 7 | 8L | L | 79 | F | NaN | 14.0 | 7 | 4.0 | 4 | 4 |
| 8 | 9L | R | 77 | M | NaN | 15.0 | 15 | 7.0 | 5 | 4 |
| 9 | 10 | L | 74 | M | NaN | 22.0 | 3 | 0.0 | 1 | 2 |
| 10 | 11WL | L | 54 | M | 28.0 | 30.0 | 0 | 0.0 | 0 | 0 |
| 11 | 12 | L | 65 | M | 26.0 | 29.0 | 1 | 0.0 | 1 | 0 |
| 12 | 13 | R | 80 | M | NaN | NaN | 3 | 0.0 | 2 | 1 |
| 13 | 14W | L | 87 | F | 16.0 | NaN | 5 | NaN | 4 | 3 |
| 14 | 15 | L | 76 | M | NaN | NaN | 4 | NaN | 3 | 3 |
| 15 | 16 | R | 18 | F | 28.0 | 30.0 | 0 | 0.0 | 1 | 1 |
| 16 | 17 | L | 61 | M | 24.0 | 27.0 | 0 | 0.0 | 2 | 0 |
| 17 | 18 | R | 72 | F | 21.0 | 21.0 | 1 | 0.0 | 1 | 2 |
| 18 | 19L | R | 52 | M | NaN | 27.0 | 21 | 13.0 | 5 | 4 |
| 19 | 20L | R | 66 | M | 22.0 | 28.0 | 7 | 3.0 | 4 | 3 |
| 20 | 21 | R | 67 | M | 18.0 | 24.0 | 5 | 1.0 | 3 | 2 |
| 21 | 22W | R | 71 | F | 11.0 | 25.0 | 2 | 1.0 | 3 | 0 |
| 22 | 23W | R | 61 | F | 25.0 | 28.0 | 4 | 1.0 | 3 | 1 |
| 23 | 24W | R | 78 | F | 15.0 | 20.0 | 19 | 7.0 | 5 | 3 |
| 24 | 25 | R | 69 | F | 27.0 | 27.0 | 0 | 1.0 | 1 | 1 |
| 25 | 26 | R | 74 | M | 23.0 | 25.0 | 0 | 1.0 | 1 | 2 |
| 26 | 27 | R | 66 | M | 23.0 | 23.0 | 0 | 0.0 | 0 | 1 |
| 27 | 28W | L | 67 | M | 28.0 | 29.0 | 0 | 0.0 | 0 | 0 |
| 28 | 29W | R | 81 | F | 18.0 | 23.0 | 1 | 0.0 | 2 | 1 |
| 29 | 30 | R | 46 | M | 29.0 | 29.0 | 0 | 0.0 | 0 | 0 |
| 30 | 31W | R | 69 | M | 24.0 | 27.0 | 1 | 0.0 | 1 | 0 |
| 31 | 32 | L | 53 | M | NaN | 27.0 | 0 | 0.0 | 1 | 1 |
| 32 | 33W | R | 57 | M | 26.0 | 26.0 | 1 | 0.0 | 1 | 0 |
| 33 | 34WL | R | 72 | F | 15.0 | 14.0 | 2 | 1.0 | 3 | 2 |
| 34 | 35 | R | 69 | M | 26.0 | 27.0 | 0 | 0.0 | 2 | 0 |
Обратите внимание, как библиотека pandas интеллектуально распознала текст „NA“ (Not Available) как отсутствующие значения и вставила в эти места NaN (Not a Number).
MoCA (Montreal Cognitive Assessment) - это тест для оценки когнитивных способностей. Как видно из таблицы не все пациенты прошли его предварительно. Большинство прошли этот тест дважды - в начале лечения и в конце. Много тех, кто прошел тест только в конце - это возможно те, кто не смог участвовать в предварительных измерениях из-за тяжелого состояния. В любом случае мы видим, что данные неполные, и это нормально для реальных данных, собранных в медицинских учреждениях. Не все пациенты соглашаются на участие в исследованиях. Некоторые по тяжести состояния просто к этому не способны. Возможно, кого-то выписали досрочно или перевели в другое отделение.
По возрасту пациентов (колонка Age) понятно, что в основном это пожилые люди. Среди тех, кому за 70, немало со сниженными интеллектуальными способностями. Однако, под ID=16 записана 18-летняя девушка, которая, впрочем перенесла инсульт почти без осложнений.
Наборы данных для публикации как правило обезличивают. Распространение сведений о болезни может сделать пациента уязвимым в ряде случаев. Этика публикации данных о пациентах определяется понятием «врачебной тайны».
Анонимизация биомедицинских данных¶
Мероприятия по анонимизации как правило сводятся к:
замена имен на коды
сдвиг дат в пределах месяца
согласие на использование в исследовательских целях
Важным является размер выборки. Например, если в работе описывается всего пятеро пациентов с редким заболеванием в какой-то стране, и из них всего одна женщина, то знания факта, что данная женщина лежала в клинике, специализирующейся на этом заболевании, достаточно для установления идентичности личности и данных о течении болезни.
В идеале данные для одного исследования собирают с нескольких клиник примерно в одно время. Это затрудняет идентификацию.
Индивидуальная динамика¶
Попробуем воспроизвести рисунок из статьи Рис. 7.
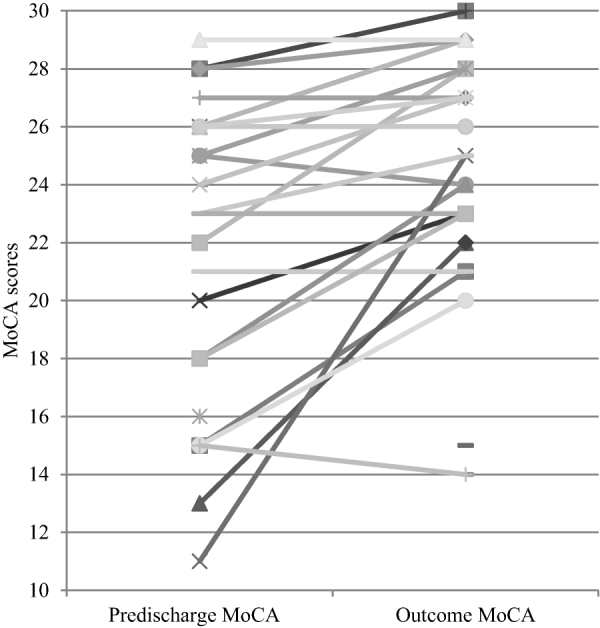
Рис. 7 График взаимодействия, показывающий изменения в оценках Montreal Cognitive Assessment (MoCA) между предварительными и окончательными измерениями.¶
Но сначала посмотрим на него и подумаем:
Есть ли в этом рисунке какое-либо применение статистических методов?
График имеет всего две координаты по горизонтальной оси: предварительный и окончательный. И значения из указанных колонок отложены по вертикальной оси. Т.е. это индивидуальные необработанные данные.
Какую полезную информацию несет этот рисунок?
Соединительные линии показывают изменения в ходе лечения. И наклон большинства линий идёт вверх - способности пациентов в общем восстанавливались. У некоторых не изменились, у двоих ухудшились, но у большинства тенденция была положительной. И это видно из этого рисунка наглядно, без использования статистики!
Как отображаются случаи, в которых одно из значений отсутствует (NaN)?
В паре мест видно маркеры, которые не соединены линиями. Это значит, что у этого значения не было пары, и соединять его линией просто не с чем.
Логика построения похожего рисунка сводится к отбору значений из соответствующих двух колонок и к подстановке к ним в качестве абсциссы значений 0.5 и 1.5. Можно и другие любые два числа, при условии установки подходящих пределов и подписей.
figsize(5,6)
xx=tile([.5,1.5],(len(D),1)).T
yy=D[['MoCA predischarge','MoCA outcome']].T
plot(xx, yy, marker='x', lw=2.5);
xlim((0,2)); xticks([.5,1.5], ['Predischarge MoCA','Outcome MoCA'])
ylabel('MoCA scores');
yticks(arange(10,32,2)); grid(axis='y', ls='-');
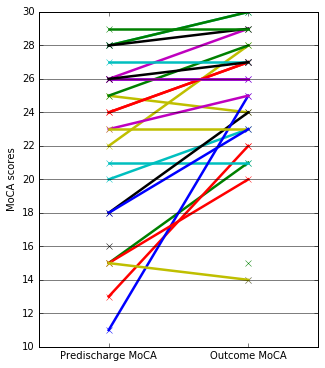
Чтобы контролировать форму и цвет маркеров - проще выводить по одной линии в цикле.
Еще проще воспользоваться готовой функцией parallel_coordinates
from pandas.plotting import parallel_coordinates
parallel_coordinates(D, class_column='ID', cols=['MoCA predischarge','MoCA outcome'], lw=2, colormap='jet');
gca().legend_.remove(); xlim(-0.5,1.5);
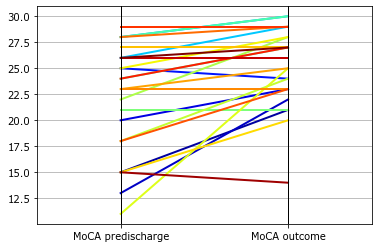
Поскольку мы извлекли данные из кода веб-страницы, то для дальнейшего анализа удобнее иметь данные в виде готового файла.
Сохраним данные в текстовый файл.
D.to_csv('stroke.tsv', sep=b'\t', index=False)
